実験データのフィッティングについて頻繁に使う機会があったので自分メモとしてまとめておきます。
フィッティングを行うにあたり、Numpy , Scipyには便利なライブラリがあります。
- Numpy :polyfit
- Scipy:optimize.leastsq
- Scipy:optimize.curve_fit
以上3つが結構有名です。簡単なフィッティングならScipy:optimize.curve_fitを使うのが一番楽だと思います。
Scipy:optimize.curve_fitを使った例
import numpy as np import scipy.optimize import matplotlib.pylab as plt x = np.linspace(0, 2*np.pi, 100) y1 = np.sin(x) y2 = 0.5*np.sin(2*x) y_noise =(y1 + y2)+ 0.08 * np.random.randn(100) plt.scatter(x,y_noise) parameter_initial = np.array([0.5, 1.0]) #a, b, c # function to fit def func(x, a, b): return np.sin(x) + a*np.sin(b*x) paramater_optimal, covariance = scipy.optimize.curve_fit(func, x, y_noise, p0=parameter_initial) print(paramater_optimal) fitting = func(x,paramater_optimal[0],paramater_optimal[1]) plt.plot(x,fitting,color="red")

このように数行でフィットまでできるので
データがそろっていてモデルがある程度絞れているならこの方法が一番早いです。
ですがちょっと複雑なことしようとするとlmfitに軍配が上がるかもしれません。
lmfitとは?
Non-Linear Least-Squares Minimization and Curve-Fitting for Pythonってサブタイトルがついてる通り非線形最小二乗法を用いたモデルフィットのためのライブラリです。
scipy.optimizeの多くの最適化方法を基にして拡張したものでLevenberg-Marquardt methodの移植からプロジェクトが始まり様々な追加がなされています。
ざっと列挙すると以下があげられる。
lmfitを使ってフィッティングを行う
lmfitの導入ですがpipなら簡単にできます。
pip install lmfit
その他Scipy,Numpy,matplotlibなんかが必要だった気がするのでご自分に合った環境づくりをしてください。
サンプルコード
import os import math import random import numpy as np import pandas as pd import matplotlib.pyplot as plt from scipy import optimize, signal from lmfit import models #gaussianを定義(ノイズ入りデータを作るためのダミーモデル) def gaussian(x, A, μ, σ): return A / (σ * math.sqrt(2 * math.pi)) * np.exp(-(x-μ)**2 / (2*σ**2)) '''fitするためのノイズ入りデータの作成''' g_0 = [250.0, 4.0, 5.0] g_1 = [20.0, -5.0, 1.0] n = 150 x = np.linspace(-10, 10, n) # ここでは2つのガウシアンモデルを足し合わせて適当なグラフを作成 y = (gaussian(x, *g_0) + gaussian(x, *g_1)) + 0.05*np.random.randn(n) fig, ax = plt.subplots() ax.scatter(x, y, s=20) '''-----------------------------------------------------------''' '''lmfitの内蔵モデルの使用''' model_1 = models.GaussianModel(prefix='m1_')#内包モデルのガウシアンを使用 model_2 = models.GaussianModel(prefix='m2_')#内包モデルのガウシアンを使用 model = model_1 + model_2#コンポジットモデルの作成 '''lmfitの内蔵モデルの初期パラメータセット''' params_1 = model_1.make_params(center=3.0, sigma=1.0)#ガウシアンのパラメータセット print(params_1) params_2 = model_2.make_params(center=-7.0, sigma=1.0)#ガウシアンのパラメータセット print(params_2) params = params_1.update(params_2)#update methodにより辞書結合 ''' updateはPythonのビルトインメソッド(もとから入ってる標準関数みたいなもの param_1とparam_2の辞書をまとめる機能がある 一般的に同名の辞書があるとその中身が上書きされてしまうがprefix='m1_'を付けているため上書きを避けることができる。 ''' output = model.fit(y, params, x=x) fig, gridspec = output.plot(data_kws={'markersize': 5})
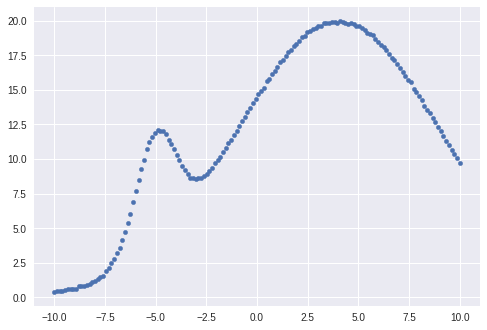
生データ
フィッティング後
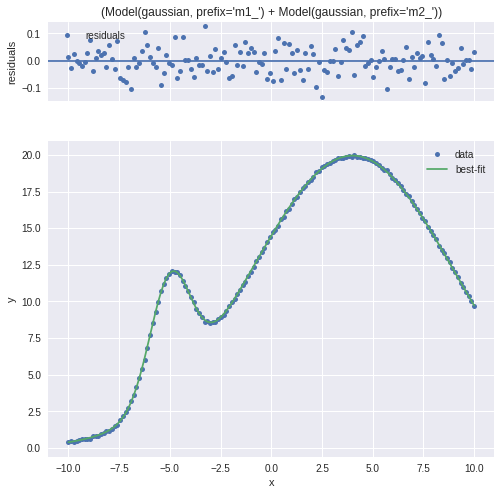
フィッティング後の残差についても出してくれます。
fit後のベストパラメータはoutput.best_valuesに格納されます。
print(output.best_values) '''-------------------------------------''' {'m2_sigma': 4.995352955273267, 'm2_center': 4.000094565660837, 'm2_amplitude': 249.88570423583715, 'm1_sigma': 1.0041137260302269, 'm1_center': -4.998780785999739, 'm1_amplitude': 20.079858838765603}
ざっと書きましたがこんなもんです。
モデル指定してモデルパラメータを設定すれば簡単にフィッティングを行うことができます。
応用的な使い方やパラメータ拘束なんかは次回書こうと思います。